预览截图
应用介绍
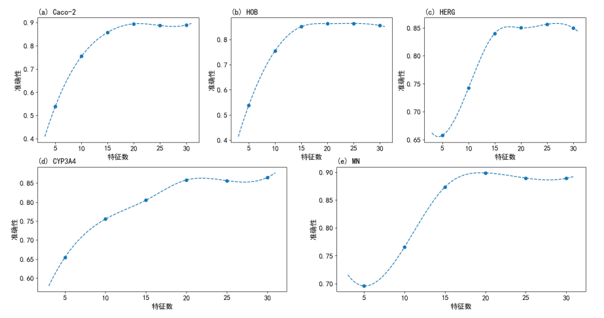
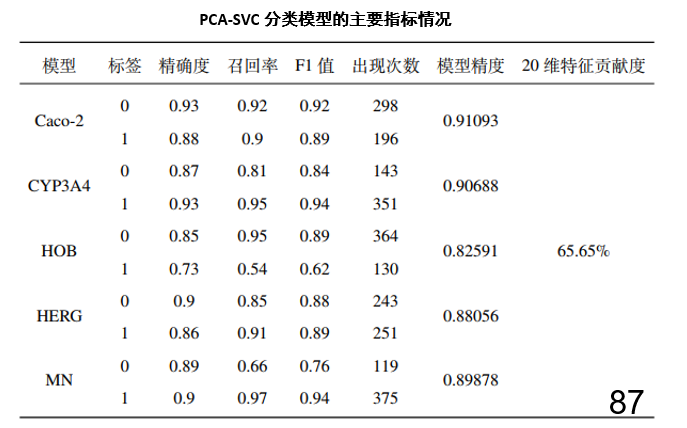
基于提供的 1974 个数据样本,对化合物的分子描述符信息、生物活性数据和 ADMET 性质数据(Absorption 吸收、Distribution 分布、Metabolism 代谢、Excretion 排泄、 Toxicity 毒性)数据进行数据挖掘和分析,建立化合物生物活性的定量预测模型、ADMET 性质的分类预测模型及整体优化模型。
©版权声明:本文内容由互联网用户自发贡献,版权归原创作者所有,本站不拥有所有权,也不承担相关法律责任。如果您发现本站中有涉嫌抄袭的内容,欢迎发送邮件至: www_apollocode_net@163.com 进行举报,并提供相关证据,一经查实,本站将立刻删除涉嫌侵权内容。
转载请注明出处: apollocode » 医学寻找病症源头的蛋白,并进行优化减少该蛋白的产生
文件列表(部分)
| 名称 | 大小 | 修改日期 |
|---|---|---|
| NSGA.py | 1.73 KB | 2021-10-18 |
| PAC+SVC(caco2).py | 1.21 KB | 2021-10-17 |
| PAC+SVC(CYP3A4) .py | 1.26 KB | 2021-10-17 |
| PAC+SVC(HERG).py | 1.25 KB | 2021-10-17 |
| PAC+SVC(HOB).py | 1.26 KB | 2021-10-17 |
| PAC+SVC(MN) .py | 1.25 KB | 2021-10-17 |
| socre(caco2).py | 0.83 KB | 2021-10-15 |
| ERα_activity.xlsx | 88.45 KB | 2021-08-12 |
| Molecular_Descriptor.xlsx | 7,995.91 KB | 2021-08-12 |
| 分子描述符含义解释.xlsx | 43.84 KB | 2021-10-14 |
| 建模.docx | 49.15 KB | 2021-10-18 |
| 抗胰腺癌候选药物的优化建模.docx | 22.51 KB | 2021-10-15 |
| 特征贡献度.py | 0.34 KB | 2021-10-16 |
| 聚类asdasd.py | 0.58 KB | 2021-10-14 |
| 获取cur_score的图.py | 0.72 KB | 2021-10-18 |
| 输出随机森林参数.py | 0.77 KB | 2021-10-17 |
| 重要性排序.py | 2.27 KB | 2021-10-17 |
| 随机森林+优化(CAO2) .py | 1.01 KB | 2021-10-18 |
| 随机森林+优化(CYP3A4.) .py | 2.36 KB | 2021-10-17 |
| 随机森林+优化(HERG.) .py | 1.01 KB | 2021-10-17 |
| 随机森林+优化(hob.).py | 2.25 KB | 2021-10-17 |
| 随机森林+优化(MN.) .py | 1.01 KB | 2021-10-17 |
| 数据 | 0.00 KB | 2021-10-20 |
| 建模代码 | 0.00 KB | 2022-06-06 |

发表评论 取消回复